人类属于一个被称为灵长类的哺乳动物类群,有趣的是,许多灵长类动物都有尾巴,这有助于它们维持身体平衡,但唯独人类和我们的近亲猿类缺乏尾巴这一身体构造。
为什么人类和猿类要把尾巴给舍弃掉?促使人类和猿类尾巴丢失进化的遗传机制究竟是什么?
2024年2月29日,美国哈佛大学/Broad研究所夏波、纽约大学朗格尼健康中心 Itai Yanai 及 Jef Boeke 团队在 Nature 期刊发表了题为:On the genetic basis of tail-loss evolution in humans and apes(人类和类人猿尾巴丢失进化的遗传基础)的研究论文【1】。该论文还被选为当期封面论文。
该研究在人类基因组中发现了一个“跳跃基因”,它可能就是导致人类尾巴消失的原因。更重要的是,这一发现还表明,人类失去尾巴的演化过程可能导致了十分危险的结果——更容易出现神经管畸形。


这一发现可以说来之不易,该论文早在2021年9月就提交给了 Nature,并在预印本平台 bioRxiv 上线,在通过一系列额外的工作开发多个基因编辑小鼠品系并进一步证明发现的基因变化具有预期效果后,最终在 Nature 上线。
许多灵长类生活在树上,为了适应树栖生活,它们发展出许多特化的结构,例如:双目向前以拥有立体视觉,方便在判断树木之间的距离;灵活的手指,能够牢牢抓住树枝;长长的尾巴,以便保持身体平衡,它们的尾巴甚至作为“第五只手”,帮助抓握树枝和攫取食物。
人类和猿类是灵长类中智慧最高的类群,而尾巴缺失是它们区别于其他灵长类最显著的解剖变化之一。人类进化研究显示,尾巴的退化促进了人类的直立行走,解放其双手促使对工具的灵活使用,这也成为人类进化的关键。然而,长久以来,促进类人猿尾巴丢失进化的遗传机制一直未被深入研究。
该论文的第一作者兼共同通讯作者夏波对《生物世界》提到,2019年的夏天,自己意外地伤到了自己的尾骨,这让自己重新想起一个小时候曾思考过的一个问题——我们人类在进化过程中是如何丢掉自己的尾巴骨的?
以此作为出发点,夏波等人分析比对了大量的灵长类基因组测序数据,并以此寻找导致类人猿尾巴消失的关键基因。据他们推断,类人猿的尾巴丢失大约发生在2500万年前,当时古人类谱系与古老的旧大陆猴子分道扬镳,只留下3-5块尾椎骨,并在后续的进化过程中形成了现代人的尾骨。
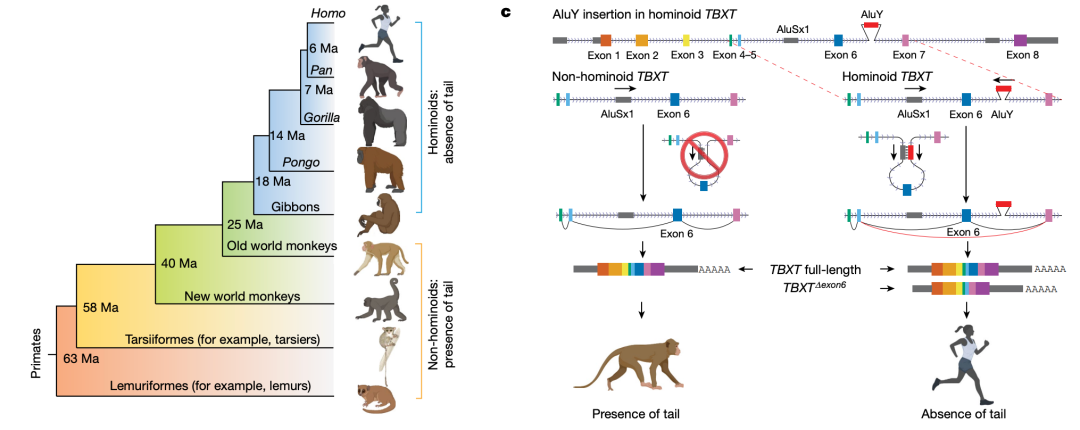
类人猿尾巴缺失的进化过程
得益于灵长类动物基因组测序项目的最新进展,使得推断基因型和表型变化之间的因果关系成为可能,并使寻找控制尾巴发育的人类特异性遗传元件成为可能。此外,对脊椎动物的发育遗传学研究已经导致了对尾巴发育基础的基因调控网络的阐明。例如,小鼠基因组信息学(MGI)数据库包括从自然突变体和与尾部表型缺失或缩短相关的诱导突变研究。
研究团队在一个名为TBXT(也叫T或者Brachyury)的基因中发现了一个强有力的怀疑对象——一个名为Alu元件的短DNA插入。有意思的是,这个Alu元件存在于所有类人猿中,但在其他灵长类动物中缺失。
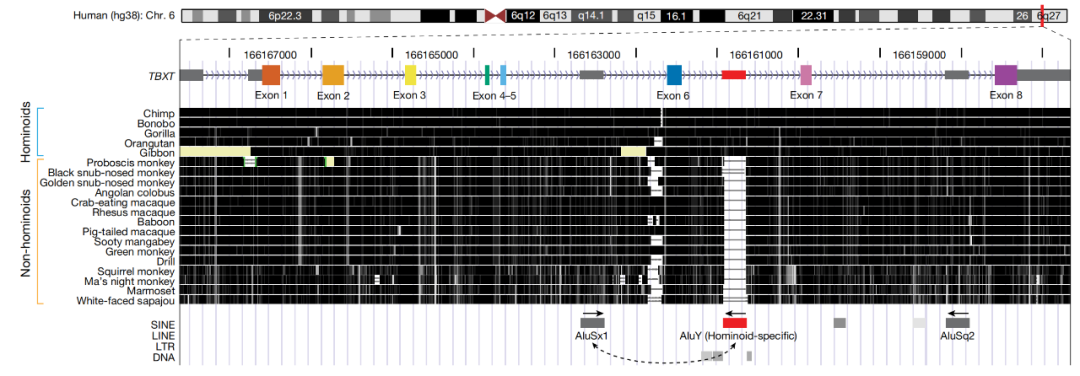
Alu元件导致TBXT的选择性剪切
Alu是人类基因组中很常见的一类可移动遗传元件(MGE),其包含超过一百万个拷贝,约占我们DNA的10%。这类被称为转座子或“跳跃基因”的DNA序列可以改变自己在基因组中的位置,它们的活动也会产生不同的结果:大部分情况下,Alu的插入不会产生任何作用;但有时候,它们会中断基因并阻止其蛋白质的产生;而在一些情况下,它们可能会改变蛋白质表达的位置或方式。
这一类人猿特异性Alu元件来自AluY亚家族,这是一种相对“年轻”的Alu,推断的插入时间与早期古人类尾巴缺失的时间点相吻合。不仅如此,夏波等还注意到,AluY存在于TBXT第6个外显子的下游,在其上游还有另一个古老的Alu元件——AluSx1,两者通过反向互补形成茎环结构,促进第6外显子的跳跃缺失,从而生成一个短的TBXT转录本——TBXTΔexon6。对此,RNA二级结构的推断模型支持了这两个Alu元件之间的相互作用。
打个形象的比喻,两个Alu元件就像衣服上的纽扣、魔术贴一样,互相结合,将一段DNA序列从基因上扯下来。
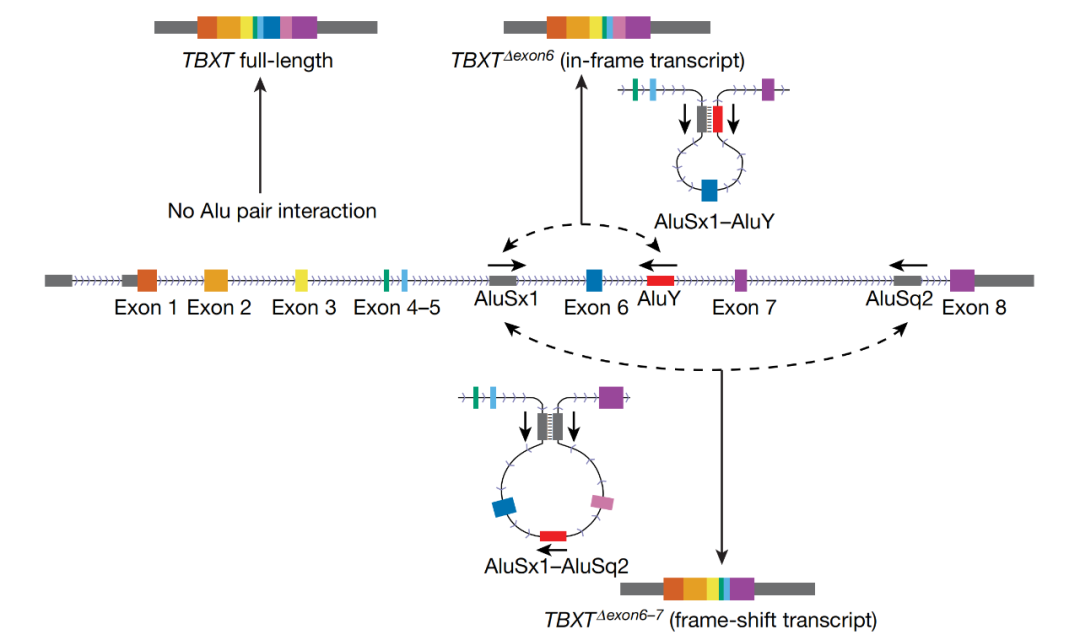
AluY和AluSx1都是诱导TBXT选择性剪接所必需
为了进一步验证AluY与AluSx1的相互作用,研究团队使用CRISPR-Cas9技术在人类胚胎干细胞中单独敲除AluY或AluSx1。他们发现,无论是删除AluY还是AluSx1,都几乎完全消除了TBXTΔexon6转录本的产生。
研究团队构建了TbxtΔexon6/+杂合子小鼠(纯合子不能存活),它们表现出强烈但异质性的尾巴形态,包括无尾和短尾表型。具体来说,63只杂合子小鼠中有21只表现出程度不一的尾巴缩短,而35只野生型同窝小鼠中没有一只表现出异常的尾巴表型。这一结果进一步证明了TBXTΔexon6转录本的存在可以导致小鼠的尾巴退化。
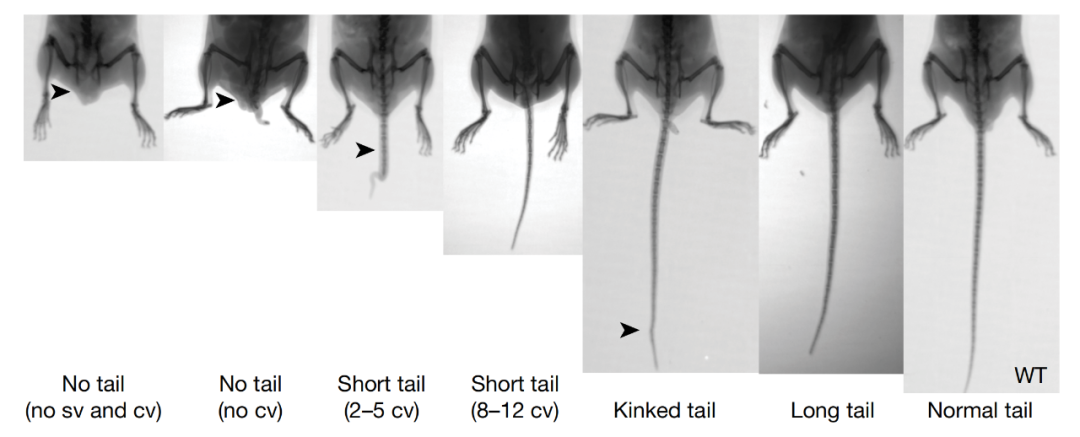
TBXTΔexon6亚型足以诱导尾巴缺失表型
不仅如此,研究团队还构建了携带人源AluY-AluSx1元件(TbxtinsASAY)的小鼠纯合子模型,虽然这些小鼠能生成TBXTΔexon6转录本,但其表达量很低,远低于人类TBXT基因的产生的TBXTΔexon6转录本,因而小鼠在表型上并没有表现出明显的尾巴缺失。
夏波等人推断是杂合子依旧能产生正常的TBXT全长转录本,只要其丰度高于某个最低阈值,或者TBXTΔexon6转录本丰度低于某个阈值,就能使尾巴表型正常。他们进一步构建了新的反向重复互补元件对(TbxtinsRCS)小鼠模型,能够以同样的可变剪切机制产生更高比例的TbxtΔexon6转录本,发现纯和TbxtinsRCS小鼠模型稳定产生更短的尾巴。
与之相印证,当生成TbxtinsRCS2/Δexon6小鼠时,TBXTΔexon6转录本表达量更高,所有19只复合杂合子小鼠均完全没有外部尾巴!这些结果使得研究团队得以最终发现Tbxt两个转录本的相对比例与尾巴长短发育的大致关系。
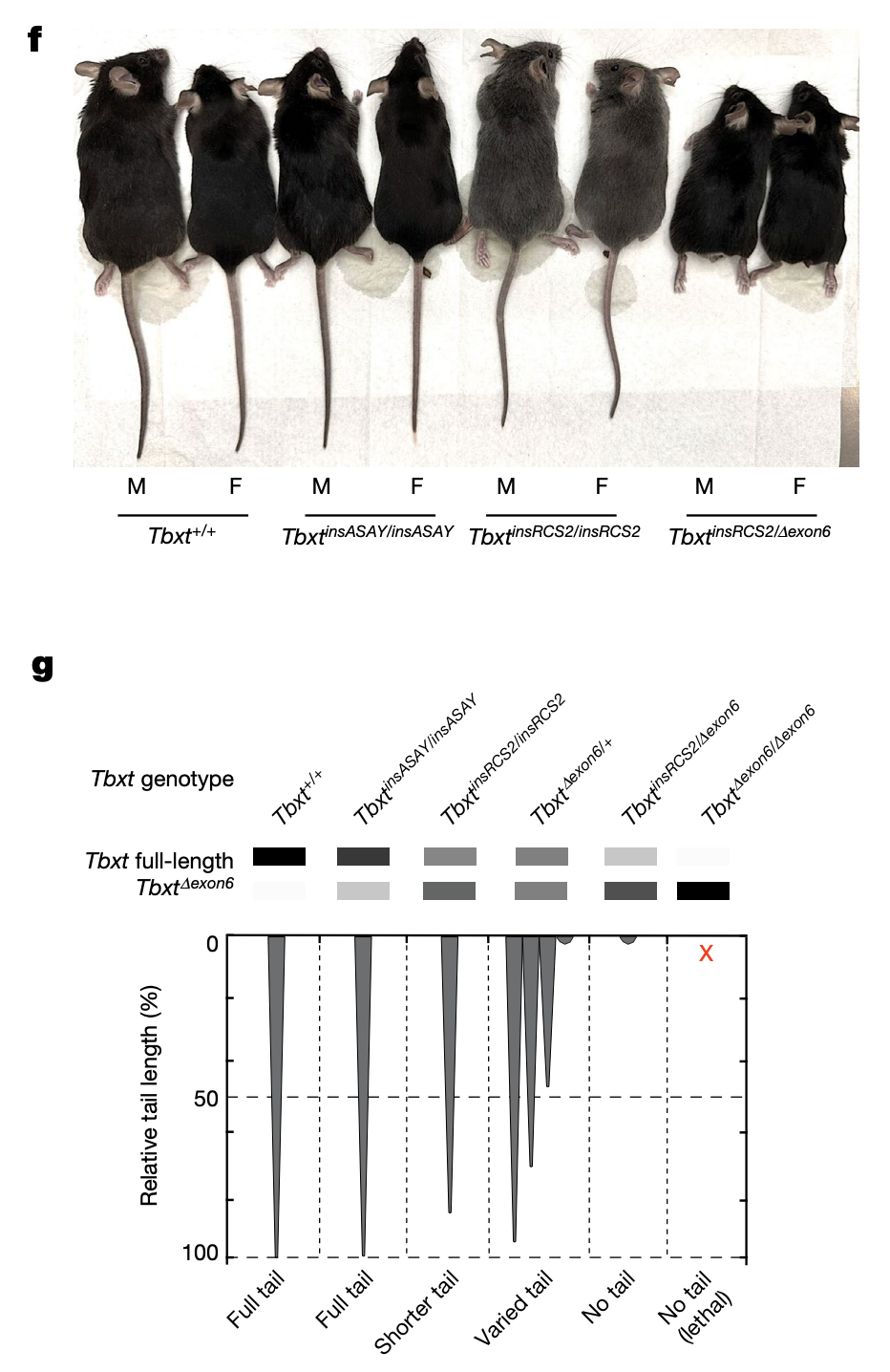
TbxtinsRCS2/Δexon6复合杂合子小鼠表现出明显的尾巴缺失表型
值得一提的是,尾骨与脊柱密切相连,那么TbxtΔexon6突变导致的尾巴缺失是否也会影响脊柱的正常发育呢?
研究团队发现,纯合的TbxtΔexon6/Δexon6突变是致命的,这些纯合子小鼠在胚胎期出现发育停滞和脊髓畸形,即便偶尔能进一步发育,小鼠也会在出生时死亡。这一现象类似于人类的神经管闭合缺陷,这类出生缺陷可能会导致脊柱裂(脊髓无法闭合),这在人类中十分常见,大约每1000名新生儿中就有1名受到影响!
事实上,Tbxt是一个保守的转录因子,TBXTΔexon6转录本可能导致一个缩短的转录因子的产生,使其与其他因子的相互作用缺失,或表现出额外的功能相互作用。这一方面的研究涉及一个复杂的基因调控网络,后续还需要更多的工作来解释两种Tbxt亚型的组合导致新的调节功能的可能性。
论文通讯作者Itai Yanai教授表示,从结果来看,人类肯定是从失去尾巴中得到了某种明显的好处,例如可以直立行走。但我们可能也为失去尾巴付出了代价——高达千分之一的神经管闭合缺陷,直到现在我们仍然能够感受到它的余威。




 联系我们
联系我们
 返回
返回