西奈山伊坎医学院的科研团队发表了一篇关于猴痘病毒基因组研究进展的文章,该研究探究了导致2023年猴痘(mpox)疫情的MPXV子支系IIb后代的基因组变化,揭示了其独特的结构特征及其变异机制。
该文章发表在《Nature Communications》上,题为“Monkeypox virus genomic accordion strategies”
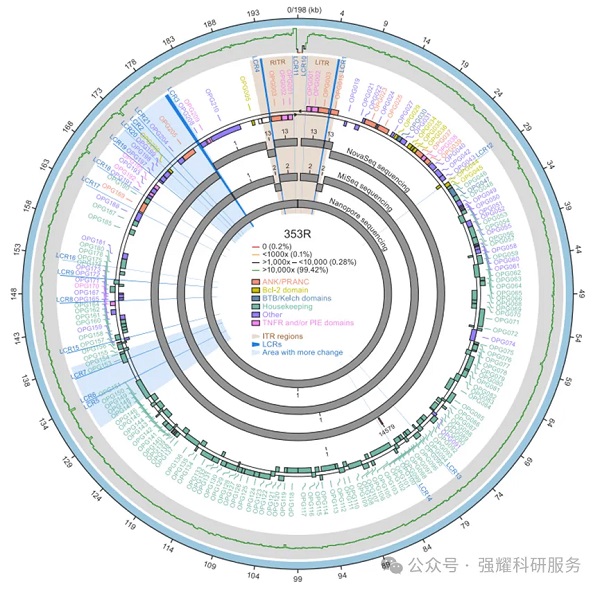
完全注释的 MPXV 分离株 353R 基因组的可视化表示
1、研究背景
猴痘病毒是一种双链DNA病毒,其基因组长度约为197kb,具有闭合的发夹状末端。基因组中包含大约193个开放阅读框(ORFs),编码至少60个氨基酸的蛋白质。这些蛋白质主要分布在基因组的中心保守区域(“核心”)和两端的终端重复区(ITRs)。其中,“核心”区域编码参与病毒转录、复制和病毒颗粒组装所需的“管家蛋白”,而两端的终端区域则编码与宿主范围和致病性相关的蛋白质。
2、研究方法与发现
基因组复杂性区域(LCRs)
研究团队通过对猴痘病毒基因组进行高通量测序,发现了一些低复杂性区域(LCRs),这些区域主要包括短串联重复序列(STRs)和同聚物(homopolymers)。LCRs广泛分布于基因组中,特别是在终端重复区(ITRs)内。这些LCRs的存在表明猴痘病毒基因组具有高度的变异性和不稳定性。

在进行宿主间序列分析时,低复杂性区域(LCR)可能比单核苷酸多态性(SNP)更能提供系统发生学信息
为了更准确地解析这些LCRs,研究者利用了单分子测序技术,对35个公开可用的猴痘病毒序列读档进行了分析。通过这种方法,他们成功解析了多个LCRs,特别是LCR3区域。结果显示,2022年疫情期间的亚支系IIb B.1序列在LCR3区域有54到62个重复单位,而2018-2019年的亚支系IIb A序列仅有12到42个重复单位。这表明LCR3区域是一个高度不稳定且易变的区域。
此外,研究还发现LCR对1/4包含一个序列重复单元[AACTAACTTATGACTT]n,在353R基因组中有16个重复单位。而在亚支系IIb B.1和A参考序列中,这一重复单位的数量分别为8个。这种差异进一步证明了LCRs在不同亚支系间的显著变化。
3、结论与未来展望
本研究不仅揭示了猴痘病毒基因组的复杂结构,还展示了LCRs在病毒变异和进化中的重要作用。这些发现对于未来的猴痘病毒监测、疫苗开发和治疗策略制定具有重要意义。未来的研究将进一步探索LCRs的功能及其在病毒生命周期中的具体作用,以期为猴痘防控提供更多的科学依据。
强耀生物可提供一系列针对猴痘病毒研究的重叠肽库,包括A35R、A29L、B6R、E8L、M1R等。欢迎您咨询订购。


 联系我们
联系我们
 返回
返回