免疫共沉淀(Co-Immunoprecipitation, CO-IP)是评估体内分子间相互作用非常重要的一个手段。对CO-IP条带的解读有助于我们去理解作者的核心创新点。但是,当我们阅读文献的时候,往往发现不同的文献,它的CO-IP条带的形式和注释是多种多样的,这不仅增加了读者的解读难度,也可能影响研究结论的可比性。本期内容将基于该痛点问题,分享一份CO-IP条带的模式图,大家可以套用简化的模式图对CO-IP条带进行分析。
一、如何解读CO-IP条带?
1. 概念理解
解读CO-IP条带需理解几个核心概念,分别是IP、IB、Input、IgG

2. 问题聚焦
本期内容集合了多篇Top期刊文献,CO-IP条带解读聚焦两个关键科学问题:
(1)第一,是否是验证分子之间存在相互作用?
(2)第二,是否是验证不同分组间分子相互作用的强弱?
3. 解读策略
针对CO-IP条带解读聚焦的科学问题,分别总结了两套解读策略:

“三步走”策略:适用于分析验证分子之间是否存在相互作用;
“四步走”策略:适用于分析验证不同分组间分子相互作用的强弱。
4. 模式图
(1)Input阳性对照组:全细胞裂解液,确保上样量一致性;
(2)IgG同型对照组:排除非特异性结合;
(3)IP阴性对照(Anti-Protein A):检测共沉淀的Protein B;
(4)分组对照:根据实验需求设置分组,比较不同分组间蛋白分子相互作用的强弱;
(5)双向互作验证(Reciprocal CO-IP):
正向实验:Anti-Protein A下拉Protein B
反向验证:Anti-Protein B下拉ProteinA
双向结果均显示特异性条带,而IgG组无信号,才能证实互作真实性。
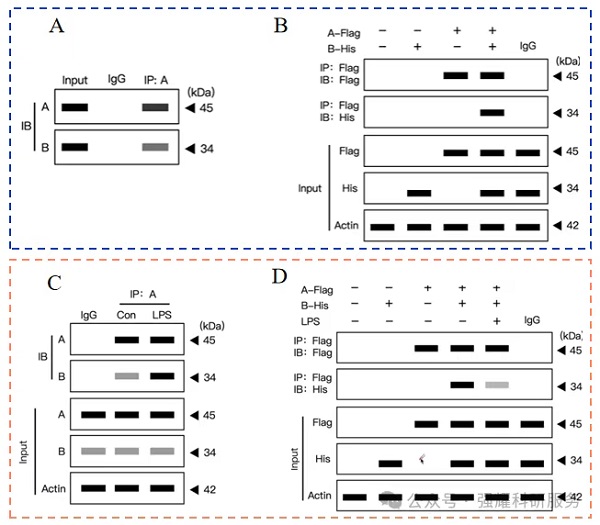
图1. 简化的CO-IP条带分析模式图
(A)为正常细胞+抗体,验证两个蛋白是否有结合;
(B)为过表达模式细胞+标签抗体,验证两个蛋白是否有结合;
(C)正常细胞+抗体,验证不同分组间分子相互作用强弱;
(D)为过表达模式细胞+标签抗体,验证不同分组间分子相互作用强弱。
二、如何证明两个蛋白是否有结合?
1. 三步走”策略

2. 两个蛋白相互作用的模式图
(1)正常细胞+抗体
为验证内源性Protein A与Protein B的相互作用,CO-IP实验严格设置对照(图2):
1、Input阳性对照组:全细胞裂解液,确保上样量一致性;
2、IgG同型对照组:排除非特异性结合;
3、IP阴性对照(Anti-Protein A):检测共沉淀的Protein B。

图2.正常蛋白相互作用模式图
(2)过表达模式细胞+标签抗体
在蛋白质相互作用CO-IP研究中,为什么要构建过表达模式细胞并结合标签系统(如Flag、His、HA等)?主要原因:
1、自然条件下,目标蛋白可能因表达量低或结合能力弱而难以被检测,过表达提高浓度,增强相互作用信号,从而能够被灵敏检测到;
2、商业化抗体易产生交叉反应或非特异结合,检测低丰度蛋白时背景干扰显著,标签抗体(如Anti-Flag、Anti-His)具有高度特异性和亲和力,可避免内源蛋白抗体的局限性;
3、目标蛋白缺乏可靠抗体,可通过融合标签(如A-Flag、B-His)实现特异检测(图3),标签系统可兼容多种检测方法(如免疫沉淀、免疫荧光),提升实验可重复性。
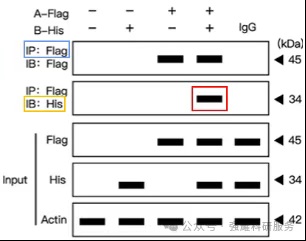
图3.标签蛋白相互作用模式图
3. 文献案例发表在《Advanced Science》, 题为“SIRT3-Mediated Deacetylation of DRP1 K711 Prevents Mitochondrial Dysfunction in Parkinson's Disease”,作者通过系统性CO-IP验证了SIRT3与DRP1蛋白之间存在相互作用(图4)。
根据“三步法”策略分析:
Step1: 找IP蛋白是什么?(DRP1蛋白)。
Step2: 找IB蛋白是什么?(DRP1、SIRT3蛋白)
Step3: 找结合条带,究竟是论证哪两个蛋白的相互作用?(DRP1作为诱饵蛋白去下拉,验证SIRT3与DRP1的相互作用)。
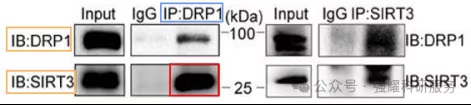
图4. 验证SIRT3与DRP1相互作用
如图4所示,与IgG同型对照相比,Anti-DRP1抗体免疫沉淀组中检测到的SIRT3蛋白信号强度显著增强。这一结果初步提示DRP1可能与SIRT3存在相互作用。为保证实验结论可靠性,需做反向下拉,使用Anti-SIRT3抗体进行免疫沉淀,检测沉淀复合物中DRP1蛋白的存在,观察与IgG组相比是否有差别。只有当双向验证(DRP1→SIRT3和SIRT3→DRP1)均获得阳性结果时,且各对照组结果符合预期标准,方可得出"SIRT3与DRP1存在相互作用"的结论。
三、如何验证不同分组结合强度?
上述是验证有或无相互作用,这部分不仅需要验证蛋白之间是否存在相互作用,还需进一步分析不同处理条件(如药物处理/时间梯度等)相互作用强度的差异。通过比较各组别结合强度的变化,可判断该疾病模型是否通过调控蛋白-蛋白相互作用强度来影响疾病发展。
1. “四步走”策略

2. 不同分组结合强度模式图
如图5所示,除原模式Input、IgG、IP、IB组别,还增设了对照组(Con )和实验组(LPS),同时目的蛋白(Protein A、Protein B)和内参蛋白(actin)都要在图中显示,以系统评估基因过表达或药物治疗对目标蛋白表达的影响。
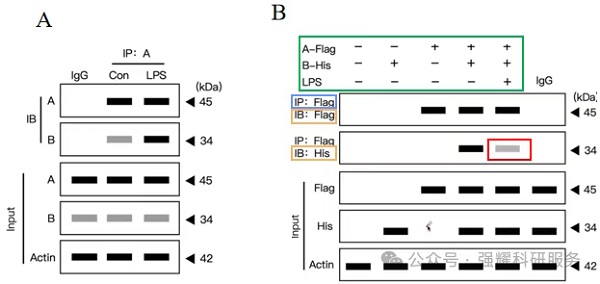
图5.不同组分结合强度模式图
在利用过表达细胞研究蛋白质相互作用时,必须从细胞定位和表达调控的角度确保实验的严谨性。针对不同表达组合(单表达/双表达)需设计系统性的对照,并明确每条条带的生物学意义。
3. 典型文献案例
不同分组相互作用包括但不限于:1、普通蛋白的相互作用;2、乙酰化蛋白相互作用;3、泛素化蛋白相互作用。以下分享三种情况的CO-IP条带解读案例。
案例一:普通蛋白结合强度
发表在《Nature Communications》题为“SKA2 regulated hyperactive secretory autophagy drives neuroinflammation-induced neurodegeneration”,作者通过CO-IP验证了SKA2与其他蛋白在不同脑区的相互作用强度(图6)。
根据“四步法”策略分析:
Step1: 找IP蛋白是什么?(SKA2蛋白)。
Step2: 找IB蛋白是什么?(SNAP29、SNAP23、STX3、SEC22B、FKBP5蛋白)。
Step3: 究竟是论证哪些蛋白间的相互作用?(SKA2作为诱饵蛋白去下拉,WB验证SKA2与SNAP29、SNAP23、STX3、SEC22B、FKBP5相互作用)。
Step4: 分哪几个组?(海马区域(HIP)、皮层区域(PFC)和杏仁核区域(AMY)。
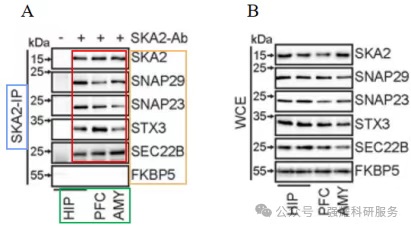
图6.验证SKA2与其他蛋白在不同脑区相互作用强度
因此,研究的是不同脑区,SKA2蛋白与不同的蛋白是否有结合,它们的结合强度怎么样?整体上来看,结合蛋白在PFC脑区表达是明显升高的,而在AMY脑区表达是明显降低的;此外STX3在PFC脑区表达最强,在其他脑区的表达都很弱(图6.A)。Input组(WCE,Whole Cell Extract)看出几个蛋白在正常情况下的分布位置及表达强弱,这看似对于证明结和强度意义不大,但从总体的严谨性来说,它是必要的(图6.B)。
案例二:泛素化蛋白结合强度
泛素化作为一种蛋白翻译后修饰作用,其实就是两个蛋白间相互作用。可以理解为靶蛋白连接到泛素蛋白,形成一个新蛋白。新蛋白具备新的功能,本质还是分子之间的相互作用(图7)。
根据“四步法”策略分析:Step1: 找IP蛋白是什么?(HA与PGAM5蛋白耦联)。
Step2: 找IB蛋白是什么?(His6与泛素蛋白UB耦联、HA)。
Step3: 究竟是论证哪些蛋白间的相互作用?(验证HA与His的结合,间接验证PGAM5与UB的相互作用)。
Step4: 分哪几个组?(K88R、K141R、K88-141R)。
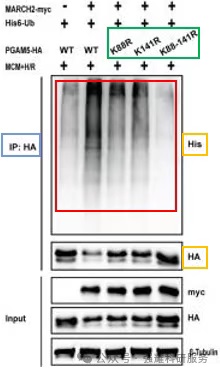
图7.验证PGAM5在不同位点的泛素化程度
从图中可以看出,在第二个泳道的的泛素化水平最明显,说明PGAM5自身可以发生泛素化。同时,通过构建不同的泛素化突变类型(K88R, K141R, K88~141R)来观察PGAM5泛素化水平的变化,可以发现,在第四泳道所指示的K88~141R突变体下,PGAM5的泛素化水平受到了明显的抑制,说明PGAM5的泛素化主要发生在其88和141位点的赖氨酸上。通过不同的分组,比较哪一个位点的泛素化会更重要,其结合强度代表着PGAM5的泛素化程度。
案例三:乙酰化蛋白结合强度
乙酰化跟泛素化的分析路径是一样的。H2O2处理后与正常组相比,乙酰化往往有改变,问题在于不知道是哪个蛋白发生了乙酰化变化,分子相互作用可以判断出哪一个蛋白它与乙酰化蛋白结合发生乙酰化作用(图8.A)。
根据“四步法”策略分析:Step1: 找IP蛋白是什么?(Flag与DRP1藕联)。
Step2: 找IB蛋白是什么?(Acetylated-Lysine、Flag、SIRT3)。
Step3: 究竟是论证哪些蛋白间的相互作用?(验证DRP1在不同分组间乙酰化程度,其次验证DRP1与SIRT3在不同分组间相互作用强度)。
Step4: 分哪几个组?(过表达DRP1-Flag、过表达DRP1-Flag+shSIRT3、过表达DRP1-Flag+SIRT3)。
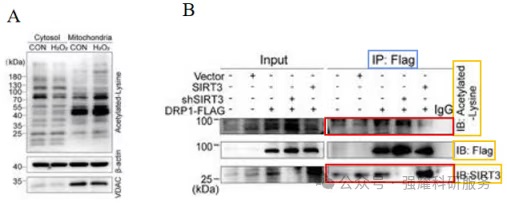
图8.验证DRPI乙酰化程度


 联系我们
联系我们
 返回
返回