11月13日,中国科学院化学研究所吴海臣研究员和高能物理研究所刘蕾副研究员等在Nature Methods期刊发表了题为:Peptide sequencing based on host–guest interaction-assisted nanopore sensing的研究论文。该研究发现,N端为苯丙氨酸的多肽与胍环分子结合后,多肽穿过α-溶血素纳米孔的阻滞时间增加,使其对特定位点的氨基酸具有极高的分辨率,可对20种氨基酸进行有效区分。在此基础上,研究者通过使用羧肽酶切割目标多肽中的氨基酸,偶联到多肽探针上,成功实现了基于纳米孔技术的多肽序列测定。

图片来源:Nature Methods
蛋白质在生物体中发挥着广泛的功能。它们由长链氨基酸组成,其序列由基因的核苷酸序列决定,然而,基因序列并不直接编码有关蛋白质丰度、翻译后修饰或剪接的信息,因此,一种高效、经济的蛋白质测序和鉴定翻译后修饰的策略是蛋白质组学研究非常需要的。
目前,Edman降解和质谱法是主要的蛋白质测序方法,但是这两种方法仍然存在很大的局限性,制约了其应用。新的蛋白质测序方法包括荧光测序、单分子肽指纹图谱、氨基酸隧道识别和基于纳米孔的蛋白质测序,其中纳米孔技术由于在DNA测序中的成功应用而具有巨大的潜力;然而蛋白质氨基酸组成种类多,肽链穿过纳米孔不均匀带电,难以控制。尽管已经报道了许多尝试用纳米孔捕获和读取蛋白质或肽链的方法,但早期的研究主要集中在观察短肽直接穿过纳米孔。科学家们尝试了多种方法解决蛋白质在穿过纳米孔前展开的问题,但仍然无法破译氨基酸序列信息。
基于此背景,吴海臣研究员和合作者提出了一种基于酶裂解和主-客体相互作用辅助纳米孔传感的多肽测序的方法。穿过α-溶血素(αHL)纳米孔的短肽迁移不产生任何可辨别电流,而当含有N端苯丙氨酸(F)的肽与胍环(CB[7])形成主客体复合物时,CB[7]穿过αHL的迁移会产生规则的、延长的电流事件。因此,研究人员设计了一系列模型肽FG1–3XD6–10,其中X表示20种蛋白氨基酸中的任何一种。
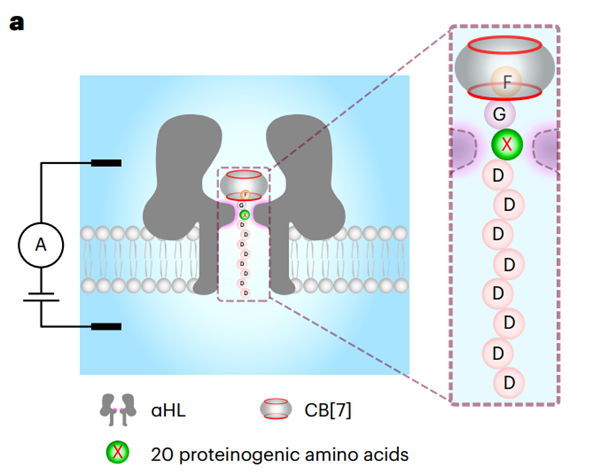
实验装置图
实验结果
1. 不同FGXD8多肽中氨基酸X的区别
研究组选用6种代表性的氨基酸:精氨酸(R)、天冬氨酸(D)、丝氨酸(S)、甘氨酸(G)、亮氨酸(L)和苯丙氨酸(F)
1)研究团队在FGXD6-10的第3个位置测试了不同长度的多D尾的影响,结果表明D8尾部是后续传感应用的最佳选择;
2)然后研究了FGXD8、FGGXD8和FGGGXD8(X代表这六种氨基酸中的一种)与CB[7]复合后的传感效果。结果显示,在多肽N末端第3个位置的情况下,这六种肽段的区分性能最好;
3)所有20种FGXD8多肽与CB[7]结合后,利用αHL突变体,研究者成功实现了对20种氨基酸的有效区分,并通过交叉验证方法证明了该策略的高效性;
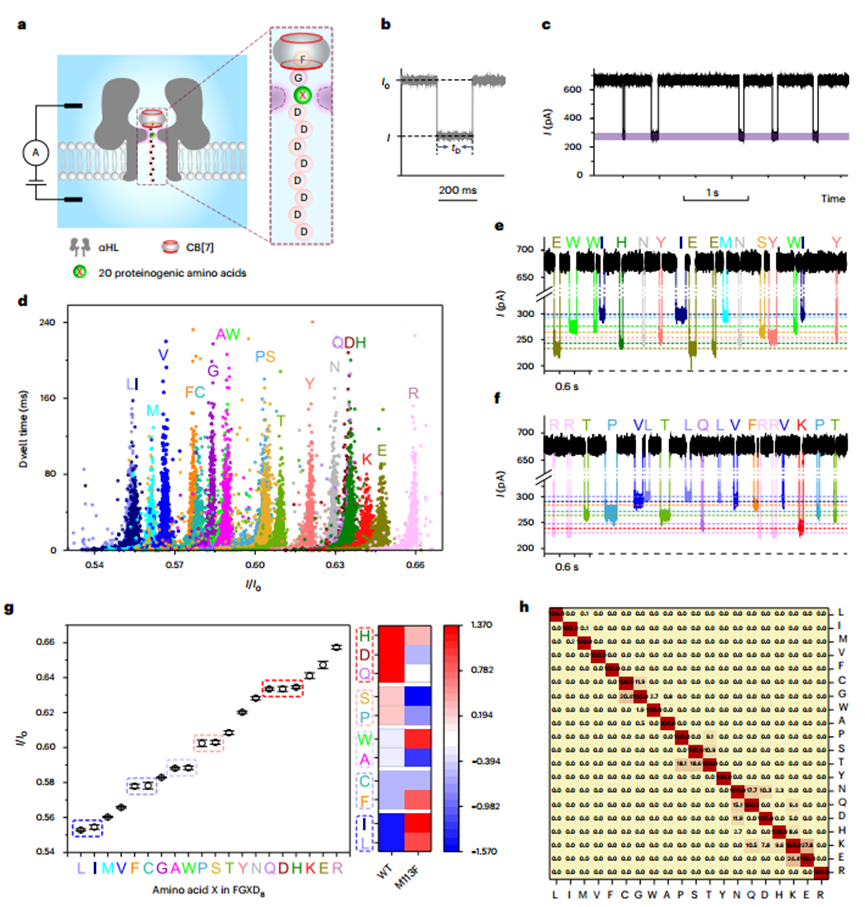
使用FGXD8探针鉴定20种蛋白质氨基酸图
4)接下来,研究者对FGXD8多肽成功区分氨基酸X的机制进行了深入研究。通过对比发现,电流阻滞与氨基酸的体积之间没有相关性,而与氨基酸的极性之间存在显著的相关性。
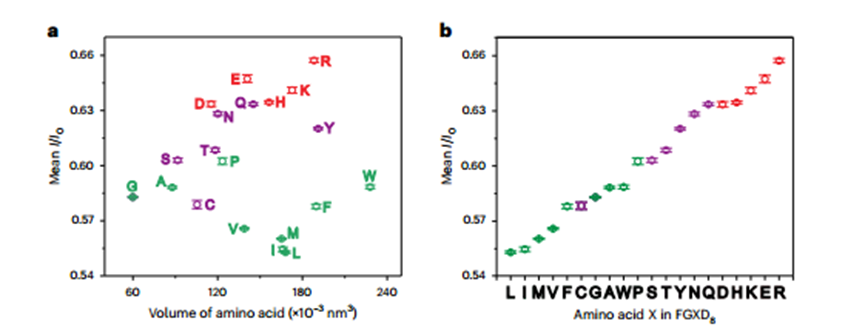
FGXD8⊂CB[7]在WT αHL中迁移的平均I/I0值与氨基酸X的性质的相关性图
2. 利用FGGCD8多肽区分氨基酸X
研究人员认为,FGXD8多肽准确区分氨基酸X的策略可用于多肽测序,该策略首先从目标多肽中切割出末端氨基酸,将释放的氨基酸共价连接到多肽探针上,然后进行纳米孔分析揭示其身份。
他们将20种氨基酸通过偶联反应分别连接到N端为苯丙氨酸、C端为8个天冬氨酸的FGGCD8多肽探针上,并通过WT α-溶血素纳米孔进行分析。虽然偶联后αHL纳米孔对20种氨基酸的分辨率略有下降,但通过利用不同的突变蛋白纳米孔,依然对全部20种氨基酸实现了有效的区分。
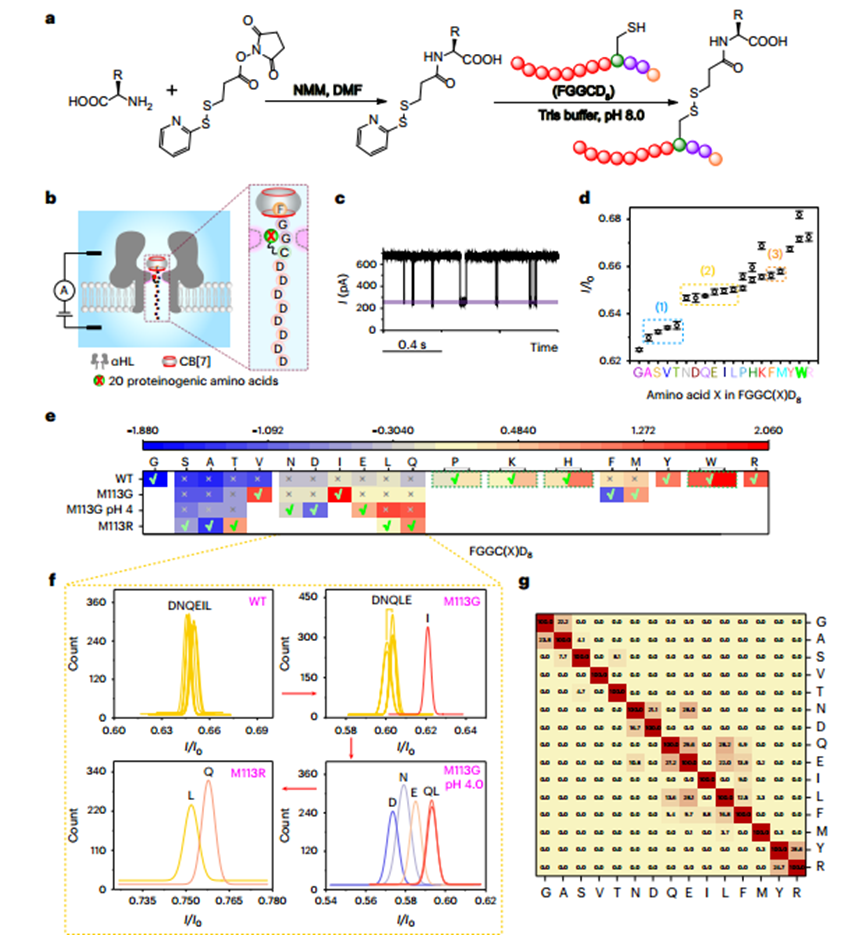
用FGGC(X)D8探针鉴定20个氨基酸图
3. 基于酶切和纳米孔技术的多肽测序验证
研究团队使用羧基肽酶A和B(CPA和CPB)来消化来自C端的肽。
1)研究者最初使用CPA和CPB的混合物裂解多肽。采用模型肽(pep1, AcRRRRRPVGLRSY;P作为终止氨基酸)经酶切消化后,从反应混合物中取样,裂解的氨基酸通过SPDP连接剂连接到FGGCD8上;最后,FGGC(X)D8肽与CB[7]复合物通过αHL纳米孔进行迁移,电流阻滞数据用于识别水解的氨基酸。然而,这种策略由于消化氨基酸的混合物的快速出现,导致了测序的不连贯性,因此研究者调整了测序策略。
2)由于裂解氨基酸在消化产物中的相对丰度随者着其在模型肽中位置的变化而变化,因此他们利用FGGC(X)D8肽段电流阻滞的相对百分比来确定模型肽段的序列。使用这种策略,设计模型肽(pep2,AcRRRRRPGAVRQF),完美揭示了其序列,与MS测序结果一致。但当消化的氨基酸数量达到8-10左右时,测序的方法会出现失调。
3)因此,为了提高测序长度,研究者设计了另一种策略,对模型肽(pep3,AcRRRRRPVRGMIYRAVQF)采用逐步酶切消化策略。他们目前正在探索这种新方法的全部潜力。
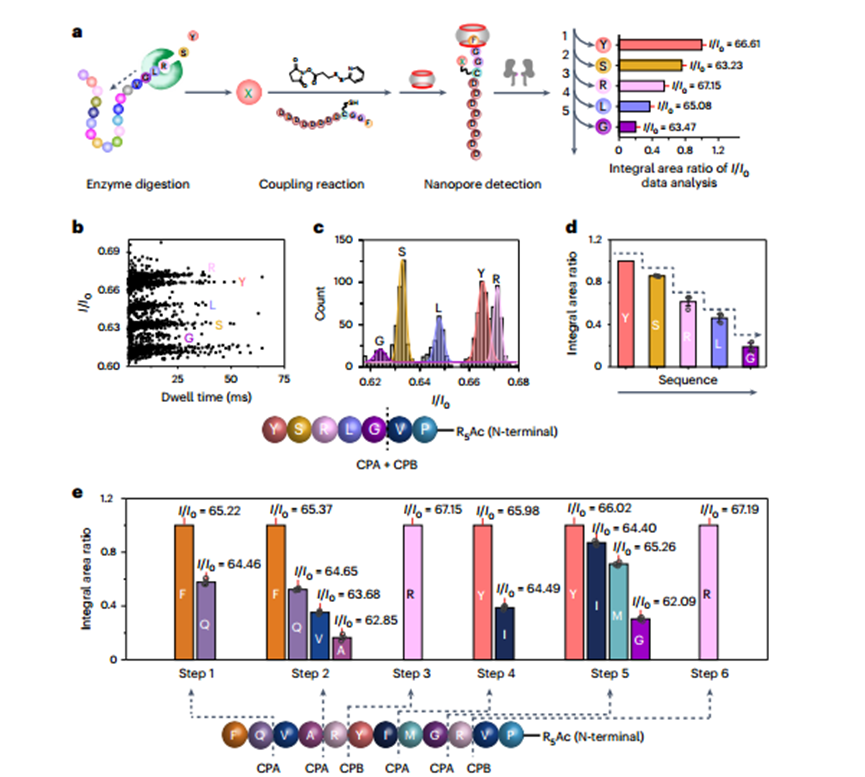
利用纳米孔进行FGGCD8的肽段测序的概念验证演示图
该项研究利用酶切和纳米孔技术,首次成功实现了多肽序列的测定。虽然还需要很多的优化和验证实验,但潜在应用前景广泛。该策略的未来发展将克服当前测序程序的局限性,为蛋白质组学研究提供一种新的蛋白质测序技术。
本篇研究中使用的所有肽均来自于强耀生物。


 联系我们
联系我们
 返回
返回